


 النبات
النبات
 الحيوان
الحيوان
 الأحياء المجهرية
الأحياء المجهرية
 علم الأمراض
علم الأمراض
 التقانة الإحيائية
التقانة الإحيائية
 التقنية الحيوية المكروبية
التقنية الحيوية المكروبية
 التقنية الحياتية النانوية
التقنية الحياتية النانوية
 علم الأجنة
علم الأجنة
 الأحياء الجزيئي
الأحياء الجزيئي
 علم وظائف الأعضاء
علم وظائف الأعضاء
 الغدد
الغدد
 المضادات الحيوية
المضادات الحيوية|
Read More
Date: 15-12-2015
Date: 19-5-2021
Date: 8-4-2021
|
Translocation Moves the Ribosome
KEY CONCEPTS
- Ribosomal translocation moves the mRNA through the ribosome by three nucleotides.
- Translocation moves deacylated tRNA into the E site and peptidyl-tRNA into the P site and empties the A site.
- The hybrid state model has translocation occurring in two stages, in which the 50S moves relative to the 30S and then the 30S moves along mRNA to restore the original conformation.
The cycle of addition of amino acids to the growing polypeptide chain is completed by translocation, when the ribosome advances three nucleotides along the mRNA. Figure 1 shows that translocation expels the uncharged tRNA from the P site, allowing the new peptidyl-tRNA to enter. The ribosome then has an empty A site ready for entry of the aminoacyl-tRNA corresponding to the next codon. As the figure shows, in bacteria the discharged tRNA is transferred from the P site to the E site (from which it is then expelled directly into the cytosol). The A and P sites straddle both the large and small subunits; the E site (in bacteria) is located largely on the 50S subunit, but has some contacts in the 30S subunit.
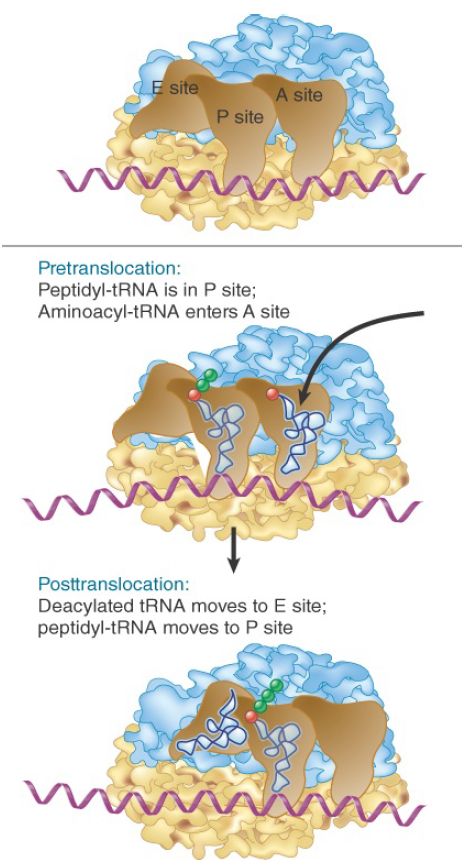
FIGURE 1 A bacterial ribosome has three tRNA-binding sites. Aminoacyl-tRNA enters the A site of a ribosome that has peptidyltRNA in the P site. Peptide bond synthesis deacylates the P site tRNA and generates peptidyl-tRNA in the A site. Translocation moves the deacylated tRNA into the E site and moves peptidyltRNA into the P site.
Evidence suggests that translocation follows the hybrid state model, which has translocation occurring in two stages. Figure 2 shows that first there is a shift of the 50S subunit relative to the 30S subunit, followed by a second shift that occurs when the 30S subunit moves along mRNA to restore the original conformation. The basis for this model was the observation that the pattern of contacts that tRNA makes with the ribosome (measured by chemical footprinting) changes in two stages. When puromycin is added to a ribosome that has an aminoacylated tRNA in the P site, the contacts of tRNA on the 50S subunit change from the P
site to the E site, but the contacts on the 30S subunit do not change. This suggests that the 50S subunit has moved to a posttransfer state, but that the 30S subunit has not moved.
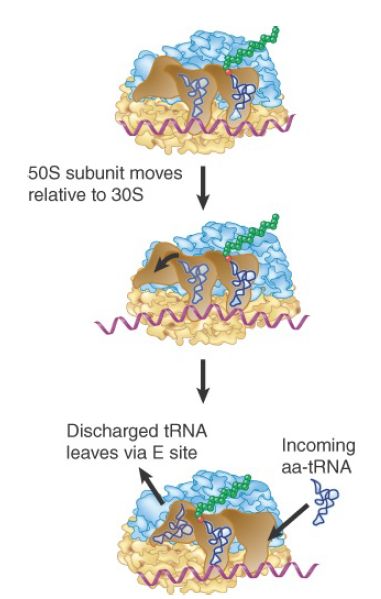
FIGURE 2. The hybrid state model for translocation involves two stages. First, at peptide bond formation the aminoacyl end of the tRNA in the A site becomes relocated in the P site. Second, the anticodon end of the tRNA becomes relocated in the P site.
The interpretation of these results is that first the aminoacyl ends of the tRNAs (located in the 50S subunit) move into the new sites (while the anticodon ends remain bound to their anticodons in the 30S subunit). At this stage, the tRNAs are effectively bound in hybrid sites, consisting of the 50S E/30S P and the 50S P/30S A sites. Then movement is extended to the 30S subunits, so that the anticodon–codon pairing region finds itself in the right site. The most likely means of creating the hybrid state is by a movement of one ribosomal subunit relative to the other so that translocation in effect involves two stages, with the normal structure of the ribosome being restored by the second stage.
The ribosome faces an interesting dilemma at translocation. It needs to break many of its contacts with tRNA in order to allow movement. However, at the same time it must maintain pairing between tRNA and the anticodon, breaking the pairing of the deacylated tRNA only at the right moment. One likely possibility is that the ribosome switches between alternative, discrete conformations, essentially acting as a Brownian motor. The switch could consist of changes in rRNA base pairing. The accuracy of translation is influenced by certain mutations that influence alternative base-pairing arrangements. The most likely interpretation is that the effect is mediated by the strengths of the alternative ribosome conformations in binding to tRNA, with elongation factors acting to stabilize certain conformations.

|
|
|
|
دراسة يابانية لتقليل مخاطر أمراض المواليد منخفضي الوزن
|
|
|
|
|
|
|
اكتشاف أكبر مرجان في العالم قبالة سواحل جزر سليمان
|
|
|
|
|
|
|
المجمع العلمي ينظّم ندوة حوارية حول مفهوم العولمة الرقمية في بابل
|
|
|