
Splice Sites Are Read in Pairs
 المؤلف:
JOCELYN E. KREBS, ELLIOTT S. GOLDSTEIN and STEPHEN T. KILPATRICK
المؤلف:
JOCELYN E. KREBS, ELLIOTT S. GOLDSTEIN and STEPHEN T. KILPATRICK
 المصدر:
LEWIN’S GENES XII
المصدر:
LEWIN’S GENES XII
 الجزء والصفحة:
الجزء والصفحة:
 9-5-2021
9-5-2021
 2166
2166
Splice Sites Are Read in Pairs
KEY CONCEPTS
- Splicing depends only on recognition of pairs of splice sites.
- All 5′ splice sites are functionally equivalent, as are all 3′ splice sites.
- Additional conserved sequences at both 5′ and 3′ splice sites define functional splice sites among numerous other potential sites in the pre-mRNA.
A typical mammalian gene has many introns. The basic problem of pre-mRNA splicing results from the simplicity of the splice sites and is illustrated in FIGURE 1. What ensures that the correct pairs of sites are recognized and spliced together in the presence of numerous sequences that match the consensus of bona fide splice sites in the intron? The corresponding GU-AG pairs must be connected across great distances (some introns are more than 100 kb long). We can imagine two types of mechanism that might be responsible for pairing the appropriate 5′ and 3′ splice sites:
- It could be an intrinsic property of the RNA to connect the sites at the ends of a particular intron. This would require matching of specific sequences or structures, which has been seen in
certain insect genes, but this does not seem to be the case for most eukaryotic genes.
- It could be that all 5′ sites may be functionally equivalent and all 3′ sites may be similarly indistinguishable, but splicing could follow rules that ensure a 5′ site is always connected to the 3′ site that comes next in the RNA.
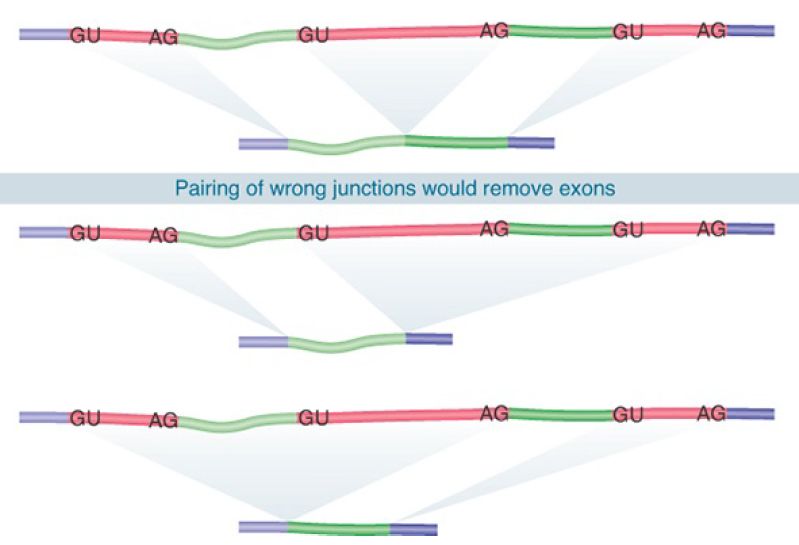
FIGURE 1. Splicing junctions are recognized only in the correct pairwise combinations.
Neither the splice sites nor the surrounding regions have any sequence complementarity, which excludes models for complementary base pairing between intron ends. Experiments using hybrid RNA precursors show that any 5′ splice site can in principle be connected to any 3′ splice site. For example, when the first exon of the early SV40 transcription unit is linked to the third exon of mouse β-globin, the hybrid intron can be excised to generate a perfect connection between the SV40 exon and the β-globin exon. Indeed, this interchangeability is the basis for the exon-trapping technique described previously in the chapter titled The Content of the Genome. Such experiments have two general interpretations:
- Splice sites are generic. They do not have specificity for individual RNA precursors and individual precursors do not convey specific information (e.g., secondary structure) that is needed for splicing. However, in some cases specific RNAbinding proteins (e.g., hnRNP A1) have been shown to promote splice-site pairing by binding to adjacent prospective splice sites.
- The apparatus for splicing is not tissue specific. An RNA can usually be properly spliced by any cell, whether or not it is usually synthesized in that cell. (Exceptions in which there are tissue-specific alternative splicing patterns are presented in the section later in this chapter titled Alternative Splicing Is a Rule, Rather Than an Exception, in Multicellular Eukaryotes.)
If all 5′ splice sites and all 3′ splice sites are similarly recognized by the splicing apparatus, what rules ensure that recognition of splice sites is restricted so that only the 5′ and 3′ sites of the same intron are spliced? Are introns removed in a specific order from a particular RNA?
Splicing is temporally coupled with transcription (e.g., many splicing events are already completed before the RNA polymerase reaches the end of the gene); as a result it is reasonable to assume that transcription provides a rough order of splicing in the 5′ to 3′ direction (something like a first-come, first-served mechanism).
Second, a functional splice site is often surrounded by a series of sequence elements that can enhance or suppress the site . Thus, sequences in both exons and introns can also function as regulatoryelements for splice-site sel ection.
We can imagine that, in order to be efficiently recognized by the splicing machinery, a functional splice site has to have the right sequence context, including specific consensus sequences and surrounding splicing-enhancing elements that are dominant over splicing-suppressing elements. These mechanisms together may ensure that splice signals are read in pairs in a relatively linear order.
 الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
 اخر الاخبار
اخر الاخبار
اخبار العتبة العباسية المقدسة


